
2019年8月5日,福建农林大学基因组中心张兴坦副教授和唐海宝教授研究组在Nature Plants杂志在线发表题为“Assembly of allele-aware, chromosomal-scale autopolyploid genomes based on Hi-C data”的研究论文,该研究利用ALLHi-C算法解决了同源多倍体基因组组装的技术难题,成功完成了同源四倍体和同源八倍体甘蔗染色体组装,安诺基因作为合作单位有幸参与了该项目的研究工作。
01
研究背景
同源多倍体在植物中较为常见,一般是由于相同的两套或多套基因组经过加倍形成的,有重要的遗传育种和农业生产价值。然而除了已发表的甘蔗割手密基因组[1]外,染色体级别的同源多倍体基因组很少被破译出来。目前Hi-C技术越来越多的应用于辅助染色体水平二倍体基因组组装,但是对于同源多倍体和近期加倍的异源多倍体来说,其同源染色体之间的Hi-C交联信号会将序列相似的等位基因片段连接在一起,导致同源染色体被错误地连接到一起,形成大量嵌合的组装,所以其组装仍存在较大困难。本研究中研究者利用ALLHi-C算法突破了同源多倍体染色体组装的技术困境,取得了开拓性的进展。
02
材料选择
同源四倍体甘蔗AP85-441,同源八倍体甘蔗Molokai-6081
03
研究结果
ALLHiC算法和验证数据集概述
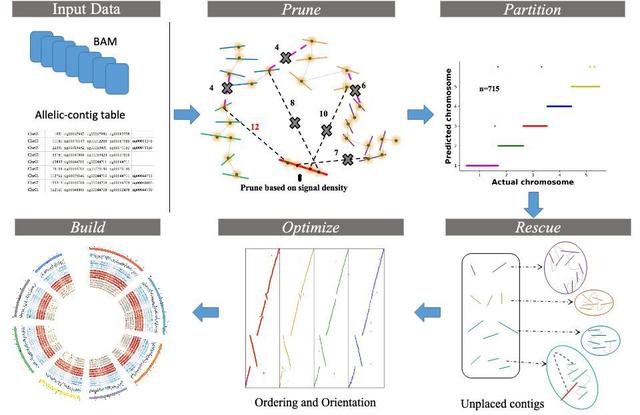
甘蔗AP85-441是通过Saccharum spontaneum花药培育的同源四倍体(1n = 4x = 32),其基因组已发表,共32条染色体,8套同源染色体,每套染色体有4个单倍型,组装出基因组大小为3.13 Gb,congtig N50为45 kb,文章以Chr4同源染色体组为例说明了ALLHiC算法在组装染色体水平同源四倍体甘蔗基因组中的应用。
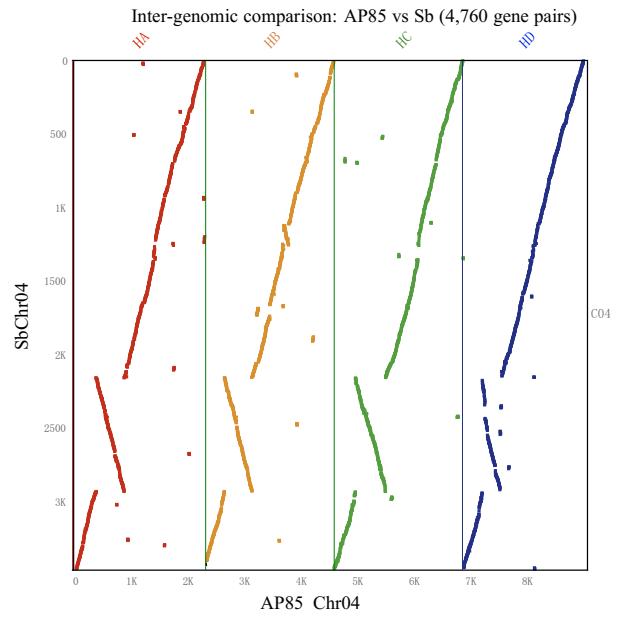
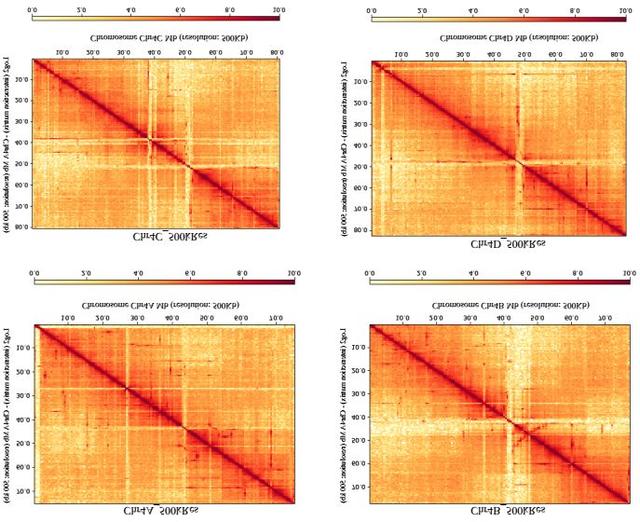
基于BLAST方法将预测的甘蔗AP85蛋白与高粱基因组比对来鉴定其等位基因contig,共8,107个注释蛋白被鉴定为2,993个等位基因contig,4,167个非冗余contig,共11,292,703条in situ Hi-C reads比对到等位基因contig上。通过修剪同源染色体Hi-C交联信号,将等位基因contig分区,优化算法进行准确排序和定向后成功组装出了相应的scaffolds。文章验证了所有scaffolds都保持了高粱和甘蔗基因组之间的高共线性,说明二者分歧时间较短,与先前遗传图谱研究结果一致。最终90.93%(3,789 / 4,167)的等位基因contig聚类组装成一组4个单倍型的同源染色体,占总contig长度的94.47%(183.85 / 194.61 Mb)。Hi-C热图显示每个同源染色体与其他染色体间没有强相互作用。以上证明运用ALLHiC算法能成功组装出染色体水平同源四倍体甘蔗基因组。
04
文章总结
ALLHiC算法一方面通过修剪Hi-C平行信号和弱信号进行等位基因分型,减少了同源染色体间的嵌合连接,另一方面通过遗传算法随机优化,极大地提高了短序列的排序和定向准确性。ALLHiC算法使多种重要多倍体基因组直接从头组装成为可能,还可用于修复已公布的多倍体物种基因组组装序列中的错误。ALLHiC算法除了适用于同源多倍体染色体组装外,同样适用于不同复杂度的基因组,包括简单的二倍体基因组、高杂合基因组和异源多倍体基因组,极大地推动了基因组领域的研究发展。
自2017年推出三代测序服务以来,尊龙凯时人生就是博先后引进了10台PacBio Sequel和4台Sequel II测序仪,产品服务类型涵盖三代基因组组装、人重测序、动植物重测序、全长转录组测序等;累计完成三代项目超800+,其中组装经验涉及中草药、林木、农作物、海洋生物、哺乳动物、昆虫和人等,并发表多篇合作文章。尊龙凯时人生就是博致力于打造专属于中国人群特有的二代+三代基因组数据库和重要农业种质资源基因库,共同推动三代测序在医学基础研究、临床转化研究和农业分子育种等领域的深度应用。
05
参考文献
[1] Zhang J, Zhang X, Tang H, et al. Allele-defined genome of the autopolyploid sugarcane Saccharum spontaneum L[J]. Nature Genetics. 2018.08.
[2] Zhang X, Zhang S, Zhao Q, et al. Assembly of allele-aware, chromosomal-scale autopolyploid genomes based on Hi-C data[J]. Nature Plants. 2019.08.
感兴趣的其他文章
岁末再添新禧|Hi-C辅助组装项目合作文章见刊Nature Communications
安诺Hi-C辅助组装合作文章再登Nature Communications
文案:产品经理 李园园