
夺嫡——同胞反目成仇的契机,国家分崩离析的原罪,宫斗作品永远的主题……不过,只关注人类兄弟间的生死争斗未免太狭隘了,同样的过程在生物界无时无刻不在上演。今天我们就透过一篇充斥硬核技术的高分文章,来近距离观看一场微观水平的“兄弟死斗”,以及由此引发的“灭国之祸”。
CTCF是一种重要的转录抑制因子,具有多个可与DNA结合的锌指结构,通过多种机制调控基因表达。CTCF最具特色的功能是通过同源二聚引起DNA成环,参与基因组拓扑结构域(TAD)边界形成,改变染色质空间结构。
作为重要的绝缘子(insulator)元件,CTCF是近年染色质结构领域热门研究对象之一,其核酸结合特征、蛋白互作关系、功能调控机制等都广受关注。中科院广州生物医药与健康研究院姚红杰研究组另辟蹊径,发现了一类可变剪接形成的CTCF异构体,并利用多种高通量测序技术深入分析了异构体的功能特征,研究成果在今年4月发表于Nature Communications。安诺基因在本研究中提供了ChIP-seq、Hi-C、RNA-seq等多种高通量测序服务,有幸为此项多组学的高水平研究添砖加瓦!


下面跟随小编先看整体思路,再读文章细节。
同根相生,兄弟同台“逐鹿”
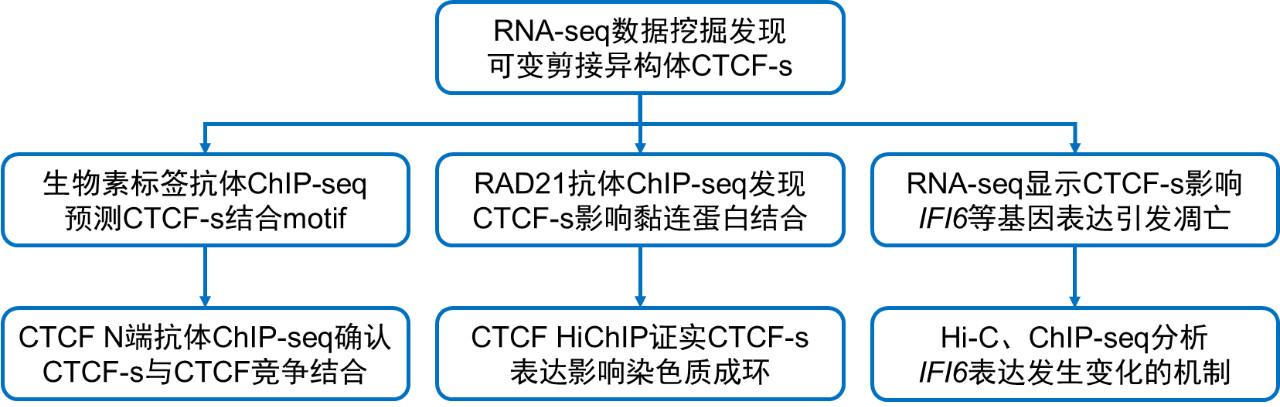
通过可变剪接,同一基因能产生结构和功能不同的RNA及蛋白质产物,构建多样化的功能调控网络。研究者在细胞系的转录组数据中发现:CTCF存在一种由可变剪接产生的异构体,并能翻译为较短的蛋白质产物。这个被命名为CTCF-s的“小弟”缺少了N端2.5个锌指(ZF)结构,具备其他8个完整ZF和C端结构域。研究者通过巢式PCR和Sanger测序证实了CTCF-s的存在,qPCR和Western blotting结果也显示CTCF-s在不同细胞系中普遍表达。
CTCF与CTCF-s的转录本构成(左)和不同细胞中的表达情况(右)
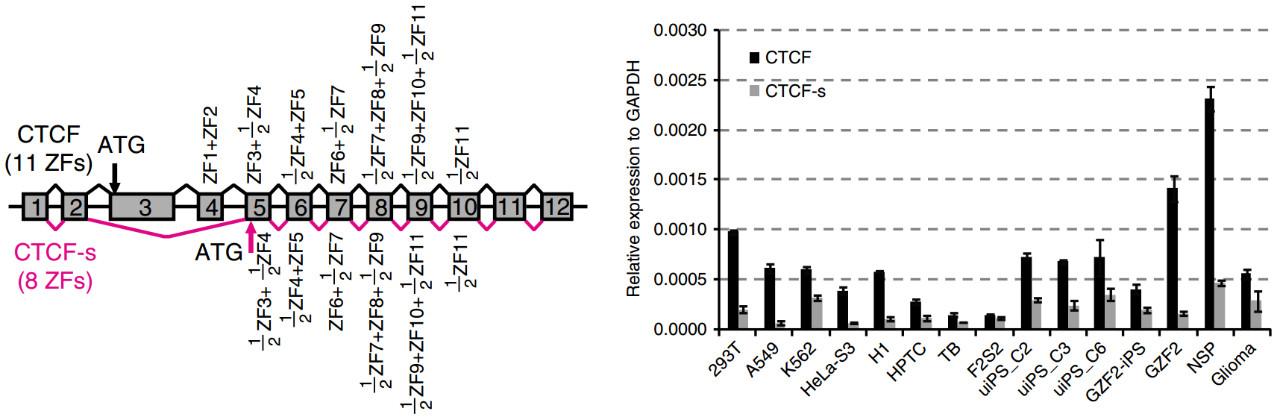
鉴于CTCF-s与CTCF的ZF结构“大同小异”,其与染色质的结合模式自然成为研究的关注点。利用ChIP-seq对带有生物素标签的CTCF和CTCF-s结合位点进行检测显示,68.8%的CTCF-s峰与CTCF峰定位重叠,表明两者之间具有功能重叠。对结合区域motif分析发现,CTCF-s结合motif较CTCF缺少2 bp——正是其缺失的ZF3所结合的区域。
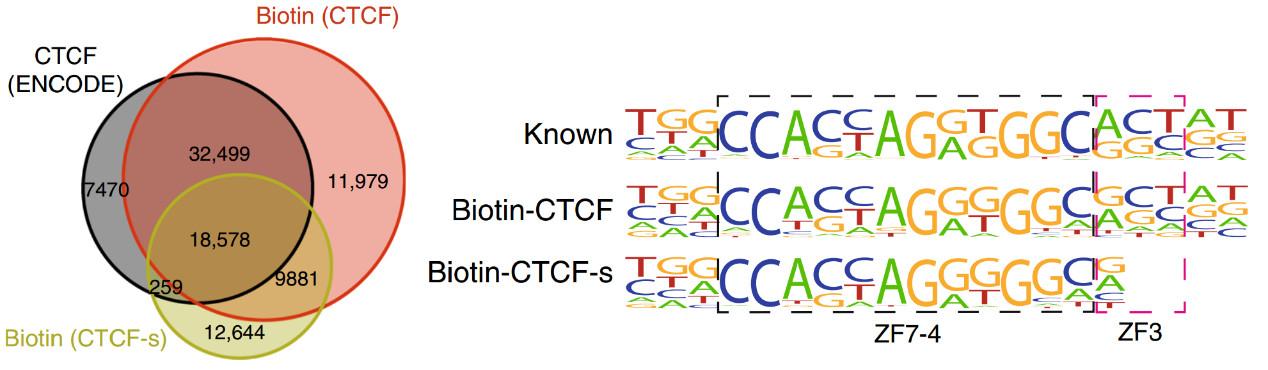
CTCF与CTCF-s结合峰的差异(左)和结合motif的变化(右)
“兄弟阋墙”,“小弟”屈居下风
CTCF和CTCF-s“兄弟俩”具有相似的DNA结合模式,在体外实验中竞争性结合DNA,细胞内情况会更复杂一些。利用CTCF N末端特异性抗体进行的ChIP-seq数据显示,在过表达CTCF-s(带FLAG标签)的细胞中,多数CTCF结合位点(下图Cluster 2)未受CTCF-s影响,大约24.2%的结合位点(下图Cluster 1)出现了显著降低的CTCF结合,这种区别与CTCF-s对应位点的结合强度有关。后续实验也证明CTCF-s与DNA的结合动力学要弱于CTCF,只能在这场“兄弟夺位战”中落于下风。
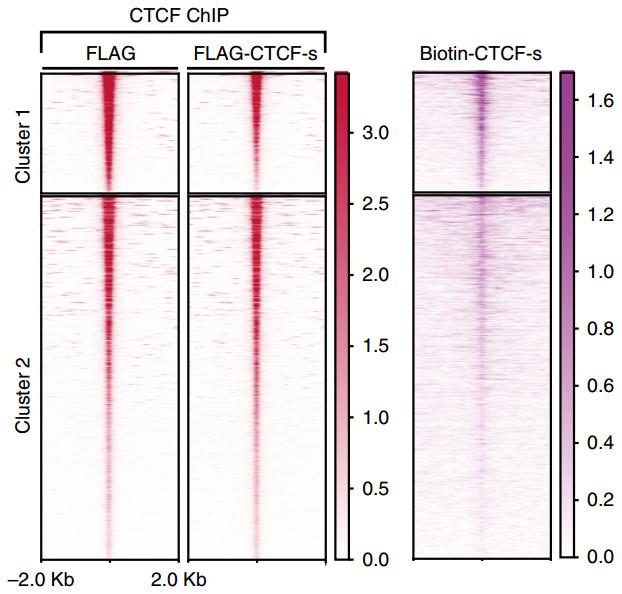
CTCF-s只在结合力较强位点(Cluster 1)上与CTCF发生明显的竞争结合
“逆袭上位”,难挽“人心离散”
考虑到CTCF对染色质空间结构的影响,CTCF-s的竞争性结合很可能对染色质成环产生影响。黏连蛋白(cohensin)复合物可以与CTCF联合搭建高级染色质构象,CTCF-s是否影响黏连蛋白与染色质正常结合是研究者首先关注的问题。对RAD21(与CTCF结合的黏连蛋白复合物核心元件)进行的ChIP-seq显示其58%的结合位点与CTCF结合位点重叠,而95.9%的对照组Cluster1位点上存在RAD21结合,可能会受到CTCF-s影响。在CTCF-s过表达细胞中,大量RAD21结合峰发生显著降低,其中多数是CTCF结合降低的位点。后续实验显示CTCF-s与RAD21的结合能力弱于CTCF,难怪RAD21对这个上位的“小弟”态度冷淡。
CTCF与黏连蛋白结合位点的减少意味着染色质成环(loop)的下调。在FLAG-CTCF-s过表达细胞中,CTCF HiChIP实验证明大量强结合的染色质loop消失,而少数新增加的loop数量和结合力都明显偏低。两端互作位点都出现CTCF结合下降的loop减少最为显著,而一端或两端都没有CTCF结合下降的loop受到影响较小,表明CTCF与CTCF-s的竞争性结合确实影响到染色质成环情况。RNA-seq结果显示,染色质loop的变化也进一步影响到启动子区有CTCF结合的下游基因表达水平。
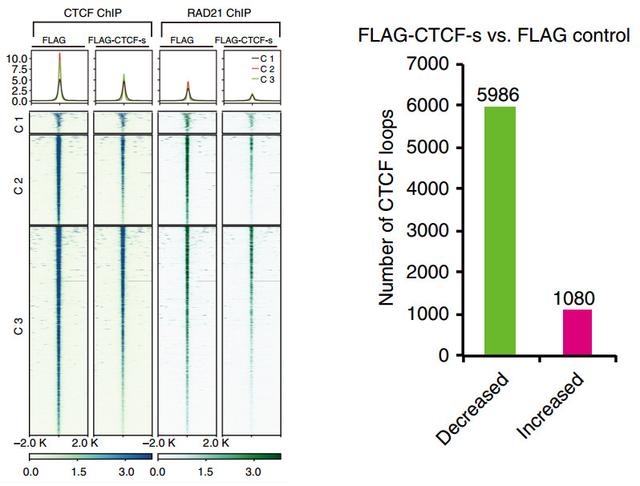
CTCF-s过表达时CTCF和RAD21结合变化(左)及染色质成环变化(右)
鸠占鹊巢,种下凋亡祸端
通过干扰染色质空间构象并影响基因表达,CTCF-s还可能产生更为深远的影响。GO分析显示CTCF-s上调的基因参与了I型干扰素信号传导和凋亡,CTCF过表达会启动细胞增殖,而CTCF-s则抑制增殖并启动细胞凋亡。干扰素诱导蛋白6(IFI6)是一种被Ⅰ型干扰素诱导上调的基因,在CTCF-s过表达细胞中发生上调,而敲低IFI6可有效阻断CTCF-s引起的细胞凋亡。
Hi-C和ChIP-seq数据显示IFI6基因位于两个相邻TAD边界附近,周围有多个会受到CTCF-s过表达影响的CTCF结合位点。通过基于启动子标记(H4K4me3)和增强子标记(H3K27ac和DNase)的ChIP-seq,研究者确定了IFI6位点周边、定位于不同sub-TAD的增强子元件分布。3C实验显示在CTCF-s表达下IFI6启动子和远端增强子发生更频繁的相互作用,表明CTCF-s通过改变IFI6周边的染色质构象和高级结构来激活IFI6基因表达。
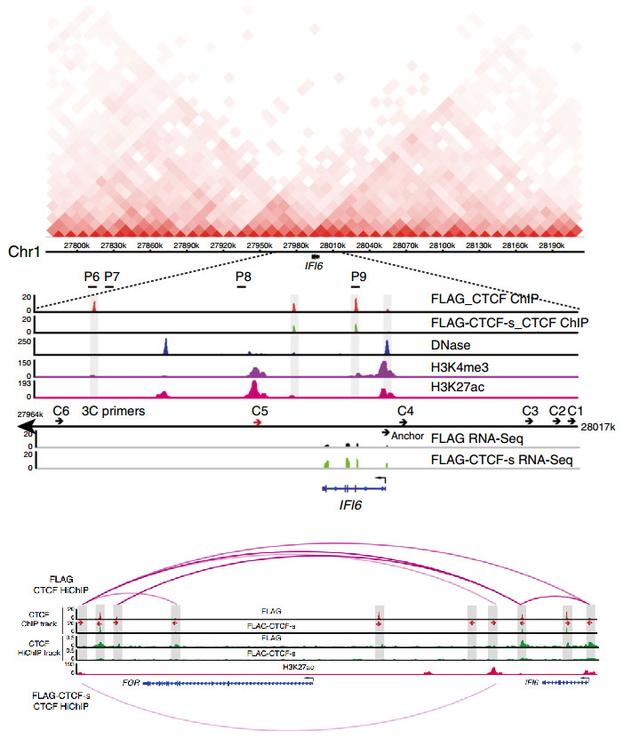
IFI6周边TAD分布(上)、功能元件分布(中)和CTCF-s过表达时的loop变化(下)
本研究从RNA-seq中发现可变剪接体CTCF-s的存在,以ChIP-seq为主、综合利用Hi-C、RNA-seq、HiChIP、ChIA-PET等高通量测序工具和其他检测技术,系统研究CTCF-s的组成、表达、DNA结合、蛋白互作等性质,展现其与CTCF竞争性结合DNA所带来的影响,并发现其具有促进细胞凋亡的功能。跟着小编读完这篇精彩的高分文章,您是否也产生了新的研究思路呢?
以蛋白-DNA互作和染色质空间结构作为对象的表观遗传研究是近年生物学研究的热点,也是不少多组学研究项目的核心。安诺基因提供包含ChIP-seq、ATAC-seq、Hi-C在内的多种表观基因组研究技术,为广大科研工作者提供优质、高效、全面的服务。赶紧与当地销售经理联系,与我们来一场多组学研究的头脑风暴吧!
参考文献:
[1] Hou C, Zhao H, Tanimoto K, et al. CTCF-dependent enhancer-blocking by alternative chromatin loop formation[J]. Proc Natl Acad Sci U S A, 2008, 105(51): 20398-403.
[2] Hashimoto H, Wang D, Horton J R, et al. Structural basis for the versatile and methylation-dependent binding of CTCF to DNA[J]. Mol Cell, 2017, 66(5): 711-720 e3.
[3] Yin M, Wang J, Wang M, et al. Molecular mechanism of directional CTCF recognition of a diverse range of genomic sites[J]. Cell Res, 2017, 27(11): 1365-1377.
[4] Li J, Huang K, Hu G, et al. An alternative CTCF isoform antagonizes canonical CTCF occupancy and changes chromatin architecture to promote apoptosis[J]. Nat Commun, 2019, 10(1): 1535.