
小科普:什么是HiFi reads?
HiFi reads(High fidelity reads)是Sequel II三代测序平台推出的兼顾长读长和高准确度的测序序列,一般采用CCS(Circular Consensus Sequencing)模式测序。在这种测序模式下,酶读长一般大于插入片段长度,因此酶会绕着模板进行滚环测序,插入片段会被多次测序。单次测序中造成的随机测序错误,可以通过算法进行自我纠错校正,最终得到高准确度的HiFi reads。要在单次测序中得到更多的HiFi reads往往需要平衡测序的酶读长和插入片段的长度,插入片段太长会导致酶无法进行滚环测序,插入片段太短又牺牲了三代长读长测序的优势。因此HiFi模式测序对酶试剂和建库过程的均一性要求较高。做完科普了,小编先带大家看一下安诺近期下机的HiFi测序数据~
安诺HiFi reads数据测评
根据前期的官方经验推荐,目前HiFi文库构建的插入片段一般为8-13 kb左右。本次尊龙凯时人生就是博构建约10 kb的HiFi文库在Sequel II平台进行测序。原始下机数据单cell产出268 Gb数据,其中酶平均读长51 kb,酶读长N50 124 kb,subreads平均读长11 kb,subreads N50 13 kb。
下机数据产出统计表

进一步利用官方软件调取CCS,设置最小pass数为3,经过调取获得CCS总数据量为22.43 Gb,CCS 序列数目为172.5万条,平均长度13 kb。与下机总数据量相比,目前CCS reads的得率约为8%,并且能够兼顾reads的读长,达到平均13 kb左右,数据质量相当不错!
CCS数据产出统计表
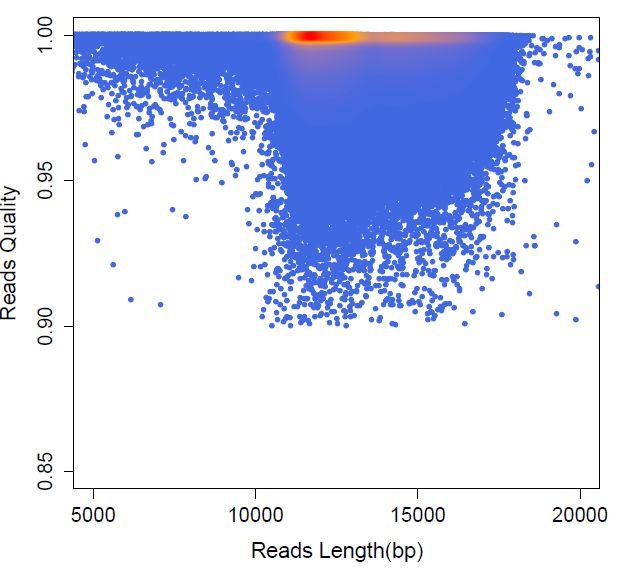
小编对我们拿到的HiFi reads进行进一步的质量评估,发现大部分HiFi reads的准确度都在0.95以上,其中约35%的reads(pass≥10)质量值达到QV30(99.9%),这样高质量的reads非常有助于研究者开展下游深入的研究。
HiFi reads有哪些用处?
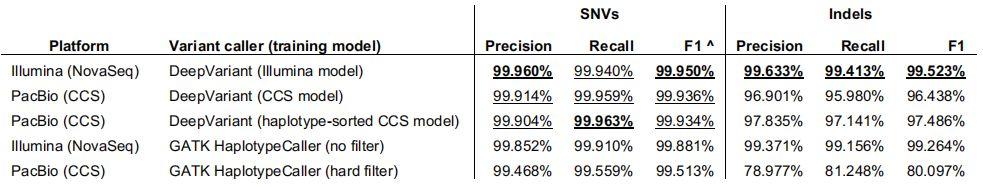
同时兼顾长读长和高准确度的HiFi reads究竟有何用处呢?小编先带大家看一篇今年发表在BioRxiv上的题为“Highly-accurate long-read sequencing improves variant detection and assembly of a human genome”的文章。在这篇文章里研究者利用约30X的CCS reads组装人基因组,通过FALCON、 Canu3和 wtdbg2等不同软件进行组装,contig N50达到15.43-28.95 Mb。从组装连续性来看,CCS reads能够做到与传统的CLR reads组装相当的结果,重要的是基因组碱基准确度得到了明显提升,基因组组装消耗的计算资源和时间大幅下降[1]。进一步利用CCS reads进行SNP、InDel等变异检测,发现CCS reads在小的变异检出率和准确度上都有显著提升,数据结果与30X的Illumina数据分析结果基本接近。
文章中CCS reads进行SNV和InDel calling统计表[1]
综上可以看出,HiFi reads无论在基因组全变异检测(SNV、InDel、SV)还是基因组de novo领域都有非常大的应用价值。目前唯一的限制因素是要获得足够的HiFi reads,测序成本的投入是比较昂贵的,但小编认为排除纯测序成本的考量,从组装计算资源节省和项目时间缩短的角度来看,HiFi reads未尝不是更好的选择。对于基因组重复序列较多的复杂基因组,目前市场上传统长读长测序准确度不高的特点给组装造成了一定的困难,高准确度的HiFi reads未来可能是一个更好的解决方案。而对于昆虫、中草药、藻类等重复序列较高、基因组较小的物种(<700 Mb),目前利用一个8 M SMRT Cell 产出的数据量基本足以支持CCS组装,性价比更高。安诺基因目前已经搭建了完善的HiFi文库建库流程和基于CCS reads组装的生信流程,期待与大家合作!
参考文献:
[1] Wenger, Pelusol, et al. Highly-accurate long-read sequencing improves variant detection and assembly of a human genome[J]. BioRxiv, 2019.