
优衣库&KAWS联名
近日优衣库和KAWS联名T恤遭“哄抢”,话题直接冲上微博热搜第二位。这两家强强联手,群众果断配合,上演一场打架、扒衣、钻门的动作大戏,这火热程度,小编只是脑补了一下就浑身发抖......对此番运作,小编看得相当通透:两家合作联名,引发爆点!
在咱们基因测序领域的创新合作火热程度不输上述场面哦~举个例子,CRISPR与Hi-C两大技术在肿瘤研究中结成同盟,开启了新的技术应用模式,接下来大家就跟随小编一探究竟~
2018年,一篇关于前列腺癌(PCa)的文章《CRISPR-mediated deletion of prostate cancer risk-associated CTCF loop anchors identifies repressive chromatin loops》发表在Genome Biology上,以非编码区风险基因作为切入点,开展前列腺癌遗传易感性的研究,非常有效地发挥了Hi-C技术探究非编码区远程调控的优势,同时基于CRISPR技术鉴定PCa细胞系中风险位点介导的CTCF loop是抑制性染色质环。接下来就顺着文章思路一步一步开始解析。

思路
前列腺癌非编码区调控
PCa是美国男性癌症死亡的第三大原因。值得注意的是,42%的PCa易感性可通过遗传因素来解释。因此,了解导致PCa潜在遗传机制至关重要。
本研究选择两种前列腺细胞(PrEC和PWPE-1)及五种前列腺癌细胞(C4-2B、PWPE-2、22Rv1、LNCap、VCap),使用Hi-C联合多组学筛选PCa风险相关的SNP位点,成功预测了一组PCa相关的非编码区SNP,并就loop染色质环影响KCNN3及KRT78基因表达机制进行深入研究,整体思路非常清晰,见图1。
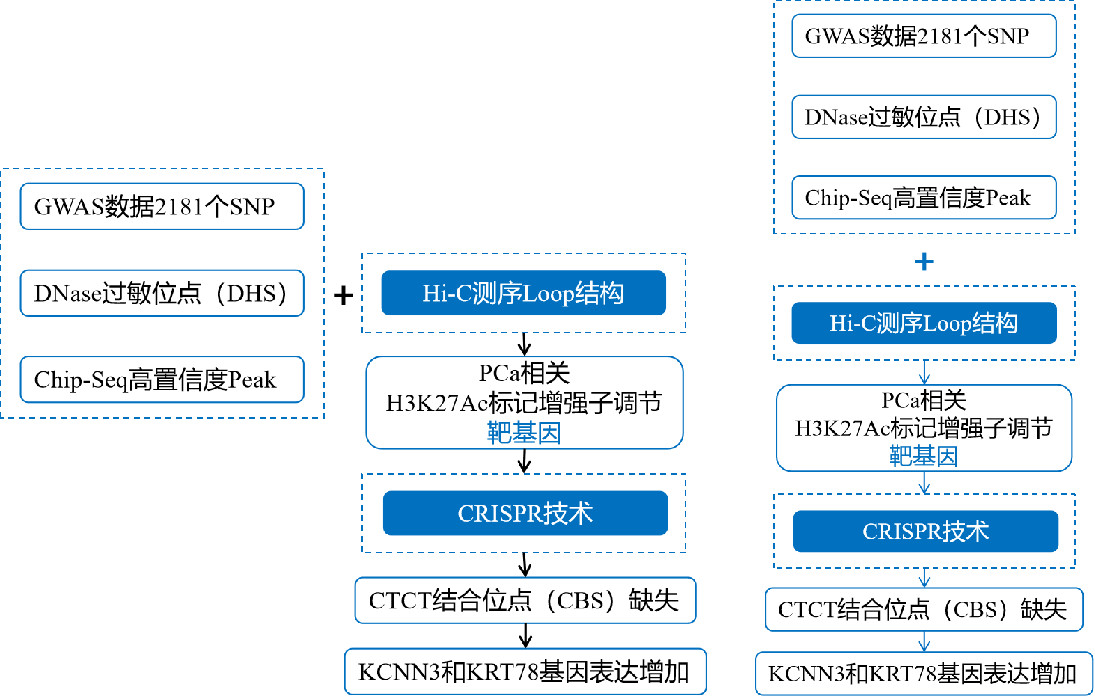
图1 实验设计流程图
筛选
Hi-C联合多组学确定PCa相关SNP
使用全基因组关联研究(GWAS)筛选人类基因组中与疾病风险相关的SNP,约100万到500万个。接下来进行精细定位研究多种族和单个大型欧洲人群,鉴定出至少2,181个PCa风险相关SNP。
因绝大多数与风险相关SNP位于基因组的非编码区域,缩小范围,使用Hi-C测序,结合Chip- seq、RNA-seq技术,按照H3K27Ac或CTCF ChIP-seq峰、高置信度(HC)峰及位于DNase过敏位点(DHS)等条件筛选,确定活性调节区域中约300个候选SNP,结果展示见图2、图3。
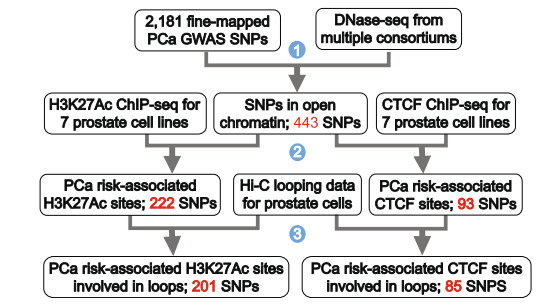
图2 鉴定染色质环中涉及PCa风险相关调节元件的实验和分析步骤
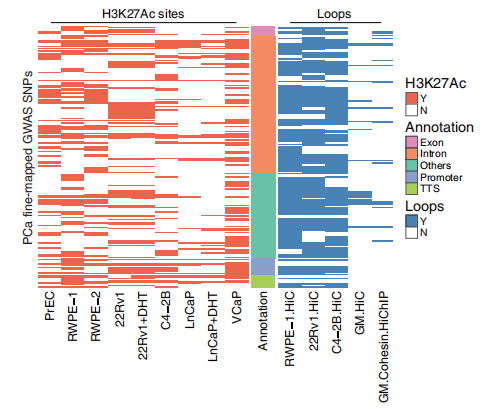
图3 与PCa风险相关H3K27Ac和loop的SNP
在选择活性调节区域中的SNP子集后,为了鉴定PCa风险相关CTCF锚定区域是否实际控制特定基因的表达,使用CRISPR / Cas9系统删除PCa风险相关的CTCF锚定区域,然后评估其影响,具体操作见图4。
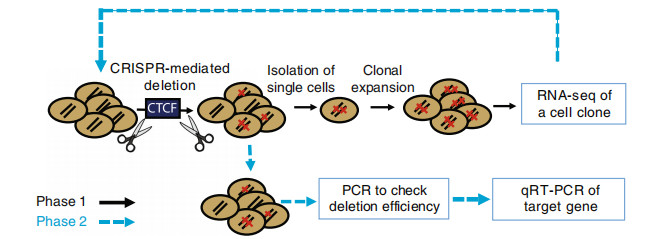
图4 与PCa风险相关CTCF结合位点基因编辑及验证流程
验证
CRISPR编辑不同CTCF结合位点
本研究选择两个PCa风险相关的CTCF锚定区域,一个在chr1上,一个在chr12上。这些区域都位于基因组的基因间区。
首先对PCa风险相关chr1 rs12144978位置的CTCF锚定区域(3个位点)开始验证,发现CTCF结合位点3单独缺失,KCNN3表达稍有增加(小于2倍),CTCF结合位点2和3同时缺失,KCNN3表达稍有增加(~7倍),而CTCF结合位点1单独缺失,则导致KCNN3表达大幅增加(几乎100倍),具体位点缺失与基因表达的关系见图5、图6。
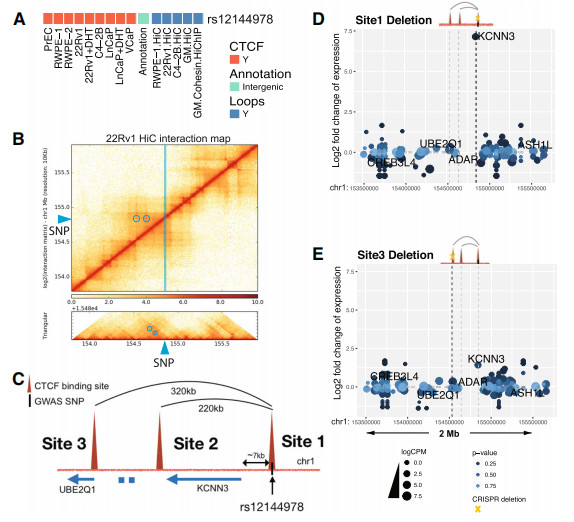
图5 KCNN3在rs12144978附近的靶向CTCF位点缺失时上调
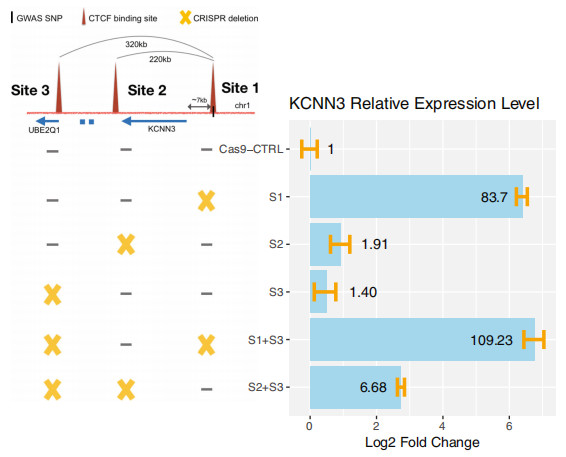
图6 rs12144978相关loop结构分析
然后对PCa风险相关chr12 rs4919742位置上的CTCF锚定区域(3个位点)进行验证,发现CTCF结合位点4单独缺失,导致KRT78、KRT4、KRT79和KRT80表达大量增加,尤其KRT78表达大幅增加(超过100倍),具体位点缺失与基因表达的关系见图7、图8。
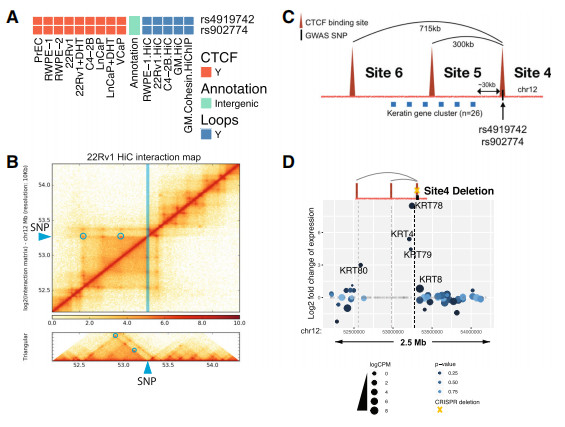
图7 KTR在rs4919742附近的靶向CTCF位点缺失时上调
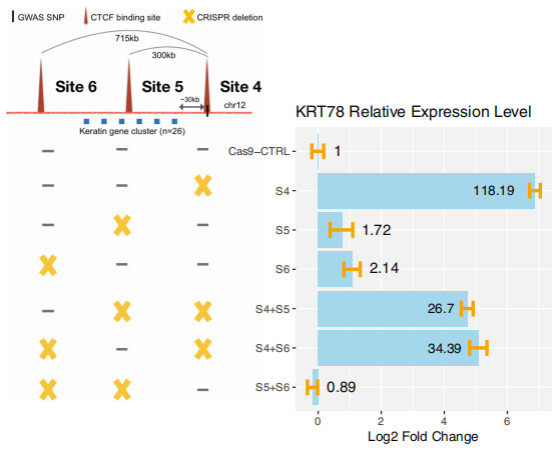
图8 rs4919742相关loop结构分析
模型
CTCF介导的环为抑制性环
通过上述研究发现,可能是PCa风险相关CTCF位点结合CTCF,阻止了KCNN3和KRT78基因的启动子与位于环外的活性增强子相互作用。为了验证这种作用模型,研究者在chr1 CTCF位点1或chr12 CTCF位点4的克隆群体中进行了H3K27Ac ChIP-seq。有趣的是,即使删除了PCa风险相关的CTCF位点,这些区域仍然是增强子荒漠区。经分析发现其可能为另一种模型,即PCa风险相关CTCF介导的环路使KCNN3和KRT78启动子与附近的活性增强子隔离,详见图9。
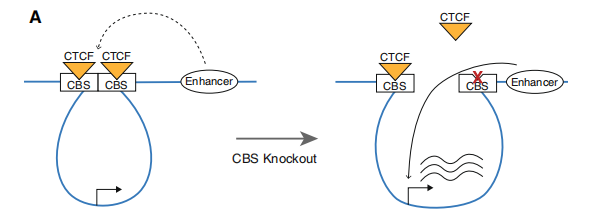
图9 PCa风险相关的CTCF环可以隔离位于环外的增强子基因
如果不是染色体构象捕获技术的诞生,也许对于非编码区的调控探索依旧是一筹莫展,如果不是基因编辑的有效验证,也许一切都只能是猜想。如今越来越多的专家学者集中肿瘤研究领域,借助Hi-C和CRISPR两大利器,深入探寻疾病发生的本质,为人类健康砥砺前行。话不多说,喜欢的话赶快收藏吧~
参考文献:
Yu Guo, Andrew A, Perez, Dennis J, et al. CRISPR-mediated deletion of prostate cancer risk-associated CTCF loop anchors identifies repressive chromatin loops[J]. Genome Biology, 2018, 19:160.
文案:Hi-C 产品经理 谷文光